Um revolucionário mecanismo de busca de DNA está acelerando a descoberta genética
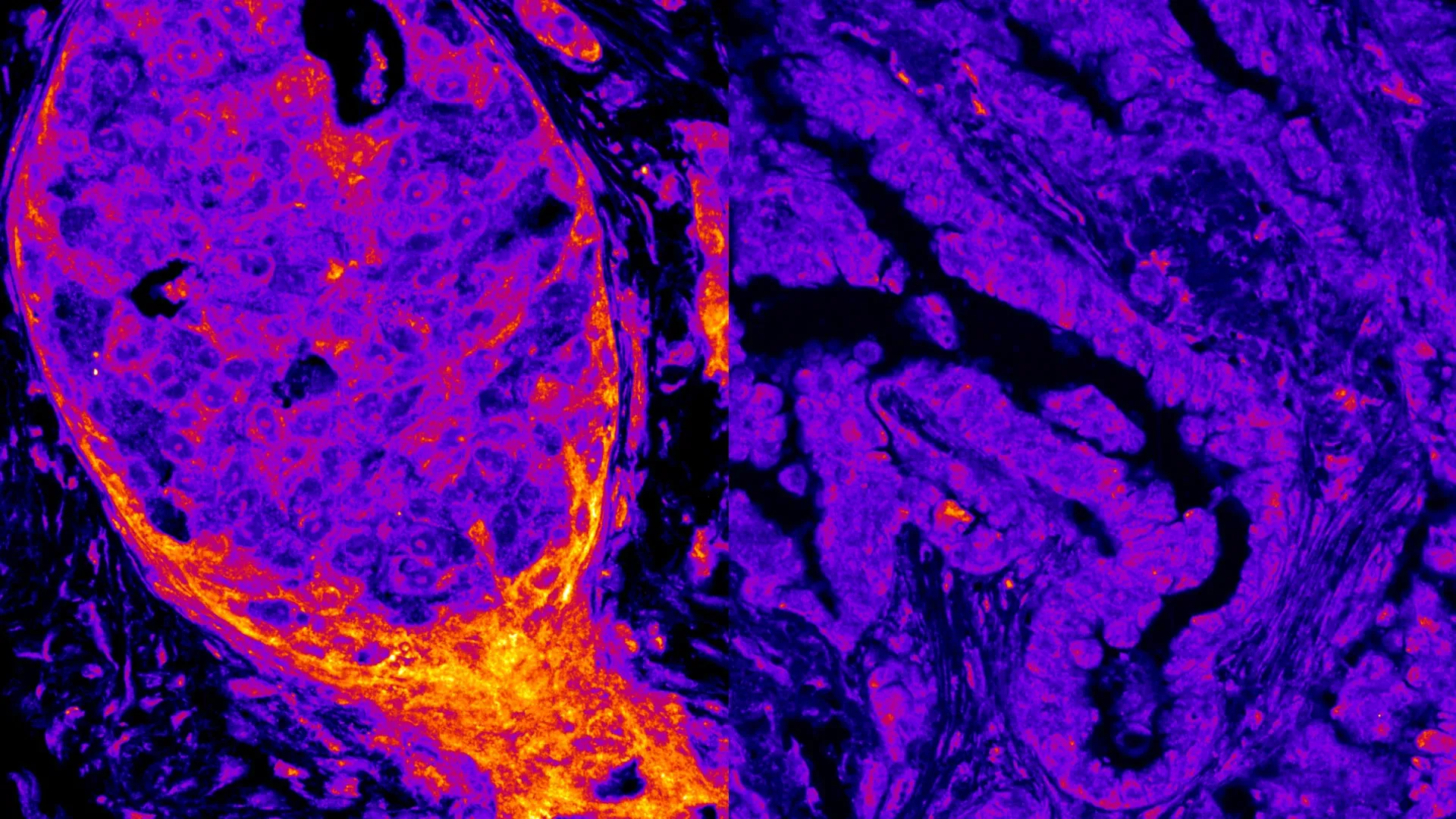
Doenças genéticas raras podem agora ser detectadas em pacientes e identificadas mutações específicas de tumores – um marco tornado possível pela sequenciação de ADN, que transformou a investigação biomédica há décadas. Nos últimos anos, a introdução de novas tecnologias de sequenciamento (sequenciamento de próxima geração) impulsionou uma onda de avanços. Durante 2020 e 2021, por exemplo, estes métodos permitiram a rápida descodificação e monitorização mundial do genoma do SARS-CoV-2.
Ao mesmo tempo, um número crescente de investigadores está a tornar os seus resultados de sequenciação acessíveis ao público. Isto levou a uma explosão de dados, armazenados em grandes bases de dados, como o SRA americano (Sequence Read Archive) e o ENA europeu (European Nucleotide Archive). Juntos, estes arquivos contêm agora cerca de 100 petabytes de informação – aproximadamente o equivalente à quantidade total de texto encontrado em toda a Internet, com um único petabyte equivalendo a um milhão de gigabytes.
Até agora, os cientistas biomédicos necessitavam de enormes recursos informáticos para pesquisar nestes vastos repositórios genéticos e compará-los com os seus próprios dados, tornando quase impossíveis pesquisas abrangentes. Pesquisadores da ETH Zurique desenvolveram agora uma maneira de superar essa limitação.
Pesquisa de texto completo em vez de baixar conjuntos de dados inteiros
A equipe criou uma ferramenta chamada MetaGraph, que agiliza e acelera drasticamente o processo. Em vez de baixar conjuntos de dados inteiros, o MetaGraph permite pesquisas diretas nos dados brutos de DNA ou RNA – como usar um mecanismo de busca na Internet. Os cientistas simplesmente inserem uma sequência genética de interesse num campo de pesquisa e, em segundos ou minutos, dependendo da consulta, podem ver onde essa sequência aparece nas bases de dados globais.
“É uma espécie de Google para DNA”, explica o professor Gunnar Rätsch, cientista de dados do Departamento de Ciência da Computação da ETH Zurique. Anteriormente, os pesquisadores só podiam pesquisar metadados descritivos e depois tinham que baixar os conjuntos de dados completos para acessar as sequências brutas. Essa abordagem foi lenta, incompleta e cara.
De acordo com os autores do estudo, o MetaGraph também é extremamente econômico. Representar todas as sequências biológicas disponíveis publicamente exigiria apenas alguns discos rígidos de computador, e consultas grandes não custariam mais do que cerca de 0,74 dólares por megabase.
Dado que o novo motor de busca de ADN é rápido e preciso, poderá acelerar significativamente a investigação – particularmente na identificação de agentes patogénicos emergentes ou na análise de factores genéticos ligados à resistência aos antibióticos. O sistema pode até ajudar a localizar vírus benéficos que destroem bactérias nocivas (bacteriófagos) escondidas nesses enormes bancos de dados.
Compressão por um fator de 300
Em seu estudo publicado em 8 de outubro em Naturezaa equipe da ETH demonstrou como funciona o MetaGraph. A ferramenta organiza e compacta dados genéticos usando gráficos matemáticos avançados que estruturam as informações de forma mais eficiente, semelhante à forma como o software de planilha organiza os valores. “Matematicamente falando, é uma matriz enorme com milhões de colunas e trilhões de linhas”, explica Rätsch.
A criação de índices para tornar pesquisáveis grandes conjuntos de dados é um conceito familiar na ciência da computação, mas a abordagem ETH se destaca pela forma como conecta dados brutos com metadados, ao mesmo tempo em que atinge uma taxa de compactação extraordinária de cerca de 300 vezes. Esta redução funciona de forma semelhante ao resumo de um livro – elimina redundâncias ao mesmo tempo que preserva a narrativa e as relações essenciais, retendo toda a informação relevante num formato muito menor.
“Estamos ultrapassando os limites do possível para manter os conjuntos de dados o mais compactos possível, sem perder as informações necessárias”, diz o Dr. André Kahles, que, como Rätsch, é membro do Grupo de Informática Biomédica da ETH Zurique. Em contraste com outras máscaras de busca de DNA atualmente em pesquisa, a abordagem dos pesquisadores da ETH é escalável. Isso significa que quanto maior a quantidade de dados consultados, menos poder computacional adicional a ferramenta requer.
Metade dos dados já está disponível agora
Introduzido pela primeira vez em 2020, o MetaGraph tem sido constantemente refinado. A ferramenta agora está acessível publicamente para pesquisas (https://metagraph.ethz.ch/search) e já indexa milhões de sequências de DNA, RNA e proteínas de vírus, bactérias, fungos, plantas, animais e humanos. Atualmente, quase metade de todos os conjuntos de dados de sequências globais disponíveis estão incluídos, e espera-se que o restante seja lançado até o final do ano. Como o MetaGraph é de código aberto, também poderia atrair o interesse de empresas farmacêuticas que gerenciam grandes volumes de dados de pesquisas internas.
Kahles até acredita que é possível que um dia o motor de busca de ADN seja utilizado por particulares: “No início, mesmo o Google não sabia exactamente para que servia um motor de busca. Se o rápido desenvolvimento na sequenciação de ADN continuar, poderá tornar-se comum identificar as plantas da sua varanda com mais precisão”.
Share this content:


Publicar comentário